Chromatin immunoprecipitation sequencing (ChIP-seq) is a flexible and powerful technique used by researchers to elucidate how gene regulation is involved with different biological events and with the progression of various conditions like cancer and neurodegenerative diseases.
When performing a ChIP-seq experiment, cross-linked DNA is first co-immunoprecipitated with the proteins it interacts with using ChIP-seq validated antibodies specific for the target protein of interest. The immuno-enriched ChIP-DNA is then used to generate a DNA library before it is sequenced using a next-generation sequencing (NGS) platform like the Illumina® HiSeq 2500 system.
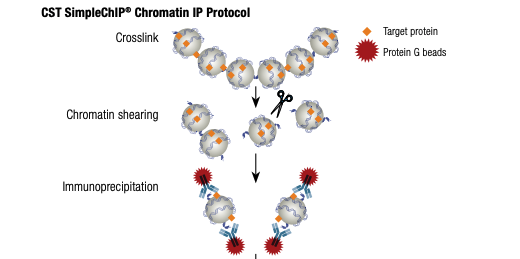
This approach gives researchers the ability to study DNA-protein interactions in an unbiased, genomic-wide manner: providing insight into disease-associated transcriptional regulation, tissue-specific epigenetic regulation, and chromatin organization (1). ChIP-seq has been used to discover new regulatory elements, like the transcription factors that make up the “enhanceosomes” that play a key role in embryonic stem cell differentiation (2). It has also revealed insights into how histone modification and DNA methylation affect chromatin dynamics, which is often dysregulated in different disease states (3-5).
Generating high-quality, reliable ChIP-seq data depends on optimizing the ChIP experiment and DNA library prep. To help you get the best possible ChIP-seq results, our new DNA Library Prep app note provides tips and recommendations from Cell Signaling Technology® (CST®) experts, including:
- How to adjust your PCR amplification protocol based on the amount of starting ChIP-DNA
- How to confirm DNA library quality before sending it off for sequencing
- How to pool DNA libraries in order to get the desired sequencing depth for each sample library
- How to quickly assess ChIP-seq data quality on a high level before starting more in-depth analysis
CST also has a comprehensive collection of protocols and resources to help with the ChIP part of your ChIP-seq experiment, as well as a complete portfolio of ChIP-seq validated antibodies, kits, and reagents.
Learn more about optimizing ChIP-seq DNA library preparation.
Learn more about ChIP and ChIP-seq.
See our full list of ChIP-seq validated antibodies.
References:
- Nakato, R. and Shirahige, K. (2017) Brief Bioinform 18(2):279-290.
- Beck, S. et al. (2015) Cell Mole Life Sci 72:199-216.
- Grosselin, K. et al. (2019) Nature Genetics 51:1060-1066.
- Schick, S. et al. (2015) J Cell Sci 128(23):4380-4394
- Arneson, D. et al. (2018) J Genet 97(3):795-806.
- Simmonds, P. et al. (2017) Genome Med 9:54.
- Berson, A. et al. (2018) Trends Neurosci 41(9):587-598.


